Travaux Pratiques #4
Algo & Prog avec R
Table des matières
1. Calcul flottant
Définissez en R la variable a dont la valeur est \(\sqrt{3}\).
- Demandez si \(a^2\) est bien égal à 3.
Expliquez. Indice :
cat(sprintf('%.17f\n', 0.1)).a <- sqrt(3) b <- a**2 cat('Est-ce que racine de 3 au carré est égale à 3 ?', b == 3, '\n') cat('Attention à l affichage de a**2 :', b, '\n') cat(sprintf('Voici la valeur de a**2 en interne : %.17f\n',b))
Est-ce que racine de 3 au carré est égale à 3 ? FALSE Attention à l affichage de a**2 : 3 Voici la valeur de a**2 en interne : 2.99999999999999956
Programmer \(100 \times 1.0\) par additions successives (à l’aide d’une boucle) : \(100 \times 1.0 = 1.0 + 1.0 + \dots + 1.0 + 1.0\). Que constatez-vous ?
acc <- 0 i <- 0 while (i < 100) { i <- i + 1 acc <- acc + 1.0 } cat(sprintf("100 * 1.0 = %.16f\n", acc))
100 * 1.0 = 100.0000000000000000
Même question pour \(100 \times 0.1\).
acc <- 0 i <- 0 while (i < 100) { i <- i + 1 acc <- acc + 0.1 } cat(sprintf("100 * 0.1 = %.16f\n",acc))
100 * 0.1 = 9.9999999999999805
2. Formule d’Euler
Le cours de maths de seconde année vous démontrera peut-être la formule suivante : \(\sum_{i=1}^{+\infty} \frac{1}{i^2}=\frac{\pi^2}{6}\).
Écrire une fonction PiEuler(n) qui calcule une estimation de \(\pi\) à partir de la somme partielle d’ordre n : \(\sum_{i=1}^{n} \frac{1}{i^2}\).
Le code ci-dessous sert à TESTER la fonction PiEuler.
PiEuler <- function(n) { ## calcul de pi avec n termes dans la somme i = 1 ## acc represente le resultat en construction acc = 1 while (i < n) { i = i + 1 acc = acc + 1/(i*i) } return(sqrt(6 * acc)) }
cat(sprintf('Calcul approximatif de pi=%.16f :\n',pi)) k <- 1 while (k <= 7){ n <- 10**k cat(sprintf("PiEuler(%d) --> %.10f\n",n, PiEuler(n))) k <- k+1 }
Calcul approximatif de pi=3.1415926535897931 : PiEuler(10) --> 3.0493616360 PiEuler(100) --> 3.1320765318 PiEuler(1000) --> 3.1406380562 PiEuler(10000) --> 3.1414971639 PiEuler(100000) --> 3.1415831043 PiEuler(1000000) --> 3.1415916987 PiEuler(10000000) --> 3.1415925581
3. Génétique des populations
Un gène donné peut exister en plusieurs allèles, avec des conséquences phénotypiques différentes: un exemple assez simple est celui des groupes sanguins, où le même gène existe en plusieurs allèles et dont le résultat observable est le groupe A,B, AB ou O. En génétique des populations, on s’intéresse à l’évolution temporelle de la fréquence des allèles dans une population.
On prend ici l’exemple d’une population haploïde (chaque gène n’existe qu’en une version, comme chez les bactéries), infinie, panmictique et à générations non chevauchantes. Si on prend un gène présent en 2 allèles A et B, on va noter \(p_t\) la fréquence de l’allèle A à une génération \(t\) (donc la fréquence de B à la même génération est \(1-p_t\)). Pour mesurer la transmission des deux allèles, on leur attribue des valeurs de sélection \(w_A\) et \(w_B\). Le cours de génétique des populations nous dit que la fréquence de l’allèle A à la génération \(t+1\) est alors:
\[ p_{t+1} = \frac{p_tw_A}{p_tw_A + (1-p_t)w_B} \]
On part de la situation suivante: \(p_0 = 0.1\), \(w_A=1\), \(w_B=0.9\). L’allèle A a une valeur sélective plus élevée que l’allèle B.
- Écrivez une fonction
ProchaineFrequence(p, wA, wB)qui prend en arguments \(p_t\), \(w_A\) et \(w_B\), et qui renvoie \(p_{t+1}\).ProchaineFrequence <- function(p, wA, wB) { pwa <- p * wA # une variable auxiliaire qui éconimise une multiplication return(pwa / (pwa + (1-p)*wB)) }
ProchaineFrequence(0.1, 1, 0.9)
[1] 0.1098901
- Écrivez une fonction
EvolutionFrequence(n, p, wA, wB)sans résultat dont l’effet est d’afficher l’évolution de la fréquence \(p_t\) surngénérations.EvolutionFrequence <- function(n, p, wA, wB) { i <- 0 while(i <= n) { cat(sprintf('Fŕequence à la génération %d : %f\n', i, p)) i <- i + 1 if( p == 0 || p == 1) { cat('Fixation : arrêt de la simulation.\n') break } p <- ProchaineFrequence(p, wA, wB) } return(p) }
- La fréquence de A atteint-elle 1 (on parle de fixation) en 100 générations ? En 200 ?
EvolutionFrequence(3, 0.1, 1, 0.9) # avec 3 générations
Fŕequence à la génération 0 : 0.100000 Fŕequence à la génération 1 : 0.109890 Fŕequence à la génération 2 : 0.120627 Fŕequence à la génération 3 : 0.132258
- Modifier la fonction pour arréter la simulation si la fixation est atteinte.
EvolutionFrequence(3, 1, 1, 0.9)
Fŕequence à la génération 0 : 1.000000 Fixation : arrêt de la simulation.
- On veut poser la même question mais pour un avantage sélectif bien moindre; on prend \(w_B=1-10^{-12}\). D’après votre programme, \(p\) augmente-t-il au fur et à mesure des générations?
EvolutionFrequence(100, 0.1, 1, 1 - 10**(-12))
- La fréquence de A atteint-elle 1 (on parle de fixation) en 100 générations ? En 200 ?
- Résultats mathématiques
Mathématiquement, on peut montrer que si \(1 > p_t\), alors \(1 > p_{t+1}\) également, et que la fixation ne peux avoir lieu qu’au bout d’une infinité de générations (dans une population infinie, ca semble raisonnable). On montre également que si \(w_A > w_B\), alors \(p_{t+1} > p_t\). Obtenez-vous la même chose avec votre simulation ? Pourquoi?
4. Dynamique des populations
Il existe en biologie de nombreux modèles de dynamique des populations, permettant de modéliser une variété de dynamiques différentes. Si vous avez fait une L1 SV à Nice, vous les avez étudiés en version continue, sous forme d’équations différentielles; on les donne ici en version discrète, où on calcule l’évolution d’une population génération après génération.
Modèle de Malthus
Le Modèle de Malthus, dont le nom a donné naissance au malthusianisme, suppose une croissance constante avec un taux \(r\) : \[ N_{t+1} = (1+r)N_t\]
\(r\) représente la différence entre natalité et mortalité: \(r > 0\) indique un surplus de natalité ; \(r \lt 0\) un surplus de mortalité.
- Écrivez une fonction
Malthus(nT, r)qui prend en arguments \(N_t\) et \(r\) et renvoie \(N_{t+1}\).Malthus <- function(nT, r) { return(nT*(1+r)) }
Malthus(100, 0.1)
[1] 110
- Écrivez une fonction
EvolutionMalthus(n, n0, r)qui affiche l’évolution de la population \(N_0\) surngénérations, puis renvoie la population finale. Ajoutez un paramètre optionnelverbosepour activer ou désactiver l’affichage.EvolutionMalthus <- function(n, n0, r, verbose = TRUE) { i <- 0; nT <- n0 if(verbose) {cat(sprintf('Population à la génération %d : %f\n', 0, nT))} while(i < n) { nT <- Malthus(nT, r) i <- i + 1 if(verbose) {cat(sprintf('Population à la génération %d : %f\n', i, nT))} } return(nT) }
EvolutionMalthus(5, 100, 0.1)
Population à la génération 0 : 100.000000 Population à la génération 1 : 110.000000 Population à la génération 2 : 121.000000 Population à la génération 3 : 133.100000 Population à la génération 4 : 146.410000 Population à la génération 5 : 161.051000 [1] 161.051
- Écrivez une fonction
PopulationMalthus(n, n0, r)qui renvoie la population finale aprèsngénérations en partant d’une population \(N_0\) par un calcul direct (sans utiliser ni boucle ni récurrence).PopulationMalthus <- function(n, n0, r) { return(n0 * ((1+r)**n)) }
Modèle de Verhulst
Le modèle de Verhulst donnera naissance aux courbes « logistiques » que les biologistes voient si souvent. Il s’écrit comme ceci: \[ N_{t+1} = \left(1 +r\left(1-\frac{N_t}{K}\right)\right)N_t\]
\(r\) a le même sens que précédemment; l’évolution de la population est multipliée par rapport au modèle précédent par \(\left(1-\frac{N_t}{K}\right)\), avec \(K\) la capacité logistique; ce terme tend à devenir faible quand \(N_t\) s’approche de \(K\).
- Écrivez une fonction
Verhulst(nT, r, k)qui prend en arguments \(N_t\), \(r\), et \(K\) et renvoie \(N_{t+1}\).Verhulst <- function(nT, r, k) { return(nT*(1+r*(1 - nT/k))) }
Verhulst(100, 0.1, 1000)
[1] 109
- Écrivez une fonction
EvolutionVerhulst(n, n0, r, k)qui affiche l’évolution de la population \(N_0\) surngénérations, puis renvoie la population finale. Ajoutez un paramètre optionnelverbosepour activer ou désactiver l’affichage.EvolutionVerhulst <- function(n, n0, r, k, verbose = TRUE) { i <- 0; nT <- n0 if(verbose) {cat(sprintf('Population à la génération %d : %f\n', 0, nT))} while(i < n) { nT <- Verhulst(nT, r, k) i <- i + 1 if(verbose) {cat(sprintf('Population à la génération %d : %f\n', i, nT))} } return(nT) } ## Note : pour $r>2$, on entre dans un régime chaotique avec non convergence vers $K$ mais oscillation entre plusieurs valeurs...
EvolutionVerhulst(5, 100, -0.1, 1000)
Population à la génération 0 : 100.000000 Population à la génération 1 : 91.000000 Population à la génération 2 : 82.728100 Population à la génération 3 : 75.139684 Population à la génération 4 : 68.190313 Population à la génération 5 : 61.836273 [1] 61.83627
- Que se passe-t’il au bout de 100 générations avec la situation suivante: \(N_0=100\), \(K=1000\), \(r=-0.1\) ?
- Et si \(r=0.1\) ? Quelle est la limite atteinte par la population au bout d’un grand nombre de générations ? Cette limite est-elle atteinte ou simplement approchée ?
- Que se passe-t-il si \(r=2.5\) ?
- Que se passe-t’il au bout de 100 générations avec la situation suivante: \(N_0=100\), \(K=1000\), \(r=-0.1\) ?
5. Méthode des tangentes de Newton HARD
Formule de Newton
La formule de Newton vu en cours pour améliorer une approximation \(a\) de \(\sqrt{r}\) est obtenue de la manière suivante. On trace la courbe d’équation \(y = f(x) = x^2-r\) qui coupe Ox précisément en \(\sqrt{r}\). 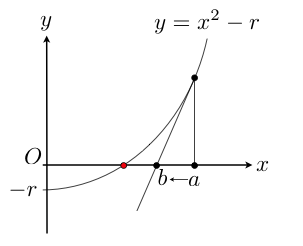
- Quelle est l’équation de la tangente (T) à la courbe au point d’abscisse \(a\) ?
- La tangente n’étant pas horizontale, calculez l’abscisse b du point d’intersection de (T) avec Ox. Vous devez retrouver la formule de Newton !
Généralisation
Les calculettes modernes possèdent souvent une touche Solve permettant de calculer une racine d’une équation \(f(x)=0\). Par exemple, il est difficile sans machine de trouver une solution réelle à l’équation \(x^5-3x+1=0\).
- Pourquoi sommes-nous sûrs qu’il y en a au moins une ?
- Nous allons faire abstraction de la fonction , la supposer dérivable et à dérivée non nulle en tout point (de sorte que la tangente à la courbe existe et n’est jamais horizontale) pour appliquer la méthode des tangentes de Newton vue ci-dessus dans un cas particulier …
On suppose que l’approximation courante est \(a > 0\). Calculez l’équation de la droite tangente à la courbe de \(f\) au point \((a,f(a))\).
- Calculez la valeur de \(b\) qui est une amélioration de \(a\).
- Ecrivez la condition pour que l’approximation courante \(a\) soit suffisamment proche de la solution. On nommera \(h\) la constante > 0 de précision.
- Ecrivez une expression mathématique utilisant \(f\), \(a\) et \(h\), qui approche la valeur de la dérivée \(f^{\prime}(a)\) de \(f\) au point \(a\).
- Programmez en R la fonction
Solve(f,a,h)retournant une approximation d’une solution defen partant de l’approximation \(a\). L’argument \(h\) gouvernera la précision.
Solve <- function(f,a,h) { ## une solution de f(x) = 0 en partant de a, et h gouverne la precision ## tant que la precision n'est pas atteinte... while (abs(f(a)) > h) { dfa = (f(a+h)-f(a))/h # approximation de f'(a) a = a - f(a) / dfa # amelioration de Newton... } return(a) # et hop ! }
g <-function(x) x**5 - 3 * x + 1 sol = Solve(g,1,0.0001) cat('x**5-3x+1 = 0 admet au moins une racine reelle car le degre est impair, ok ?\n') cat('Une solution de x**5-3x+1 = 0 :', sol, "\n") cat('Verification : g(sol) =',g(sol),'où e-05 signifie *10**(-5)\n')
x**5-3x+1 = 0 admet au moins une racine reelle car le degre est impair, ok ? Une solution de x**5-3x+1 = 0 : 1.214649 Verification : g(sol) = 1.056702e-05 où e-05 signifie *10**(-5)
Applications numériques à faire sur ordinateur
Utilisez la fonction Solve(f,a,h) pour faire afficher une valeur approchée :
- de \(\sqrt{2}\),
- puis de \(\sqrt[3]{2}\),
- puis d’une solution de l’équation \(x^5-3x+1=0\),
- puis de l’équation \(cos(x)=x\),
- et enfin du nombre \(\pi\).
cat('Approximation de la racine cubique de 2 :',Solve( function(x) x**3-2, 1, 0.0001), "\n") cat('(la "vraie" valeur est ',2**(1/3),')\n') cat('Approximation de pi comme solution de sin(x) = 0 :',Solve(sin, 3, 0.0001), "\n") cat('(la "vraie" valeur est ',pi,')\n') sol <- Solve(function(x) x-cos(x), 1, 0.0001) cat('Solution de cos(x) = x :', sol, "\n") cat('Verification de cos(',sol,') == ', cos(sol), ':', sol == cos(sol), "\n")
Approximation de la racine cubique de 2 : 1.259934 (la "vraie" valeur est 1.259921 ) Approximation de pi comme solution de sin(x) = 0 : 3.141593 (la "vraie" valeur est 3.141593 ) Solution de cos(x) = x : 0.7391132 Verification de cos( 0.7391132 ) == 0.7390663 : FALSE
N.B. Au moment d’utiliser la fonction Solve(f,a,h), il n’est pas nécessaire que la fonction f soit déjà définie. On peut passer une « fonction anonyme » en paramètre de Solve.
Par exemple, la fonction QUI N’A AUCUN NOM s’écrit en R :
function(x) x**2 – 1
On pourra donc demander par exemple
Solve((function(x) x**2 – 1), 3, 0.01)
Comparaison avec la fonction uniroot
Le langage R propose une fonction pour trouver la racine d’une fonction d’une seule variable.
f <- function(x) x**2 - 1 uniroot(f, c(0, 10))
Refaites les applications numériques avec uniroot et comparez les résultats avec ceux de votre fonction Solve.
6. Racines d’un trinôme HARD
Il est fortement conseillé de lire la page wikipedia sur les équations du second degré ou encore mieux Scilab is not naive.
Méthode naïve
Écrire une fonction triroot(a,b,c) prenant en paramètre les coefficients a, b et c d’un trinôme et renvoyant les racines réelles de l’équation \(ax^2 + bx + c =0\). Plus précisément, la fonction renvoie :
NULLsi l’équation n’admet pas de racines réelles ;- un scalaire si l’équation admet une racine double ;
un vecteur à deux éléments si l’équation admet deux racines distinctes.
triroot <- function(a, b, c) { delta <- b**2 - 4*a*c if(delta < 0) { return(NULL) } else if(delta > 0) { s <- sqrt(delta) return(c( -b + s, -b - s)/(2*a)) } else { return -b/(2*a) } }
Vérifiez votre programme en utilisant la fonction prédéfinie polyroot.
triroot(1,-3,2) polyroot(c(2,-3,1))
[1] 2 1 [1] 1+0i 2-0i
Autour de la validité de la comparaison à 0
Tester votre programme avec le code ci-dessous. Quelle conclusion en tirez-vous?
triroot(0.01,0.2,1) polyroot(c(1,0.2,0.01)) triroot(0.011025,0.21,1) polyroot(c(1,0.21,0.01025))
Proposer une amélioration permettant d’éviter le problème ci-dessus grâce à la fonction all.equal.
Autour de l’annulation massive
On va analyser l’erreur d’arrondi pendant le calcul du discriminant quand \(b^2 >> 4ac\) en étudiant le trinôme \(\epsilon x + \frac{x}{\epsilon} - \epsilon\).
li <- head(seq(0.0001,0,length.out=4),-1) for (epsilon in li) { e1 <- epsilon**2 #cat("Epsilon:", epsilon, "\n")) cat("Expected root:",e1, "\n") r1 <- triroot(epsilon, 1/epsilon, -epsilon)[1] cat("Naive method:",r1, "error=", format(abs(1-r1/e1), digits=3), "\n") r2 <- Re(polyroot(c(-epsilon,1/epsilon,epsilon))[1]) cat("R method:",r2, "error=", format(abs(1-r2/e1),digits=3), "\n") }
Expected root: 1e-08 Naive method: 9.094947e-09 error= 0.0905 R method: 1e-08 error= 0 Expected root: 4.444444e-09 Naive method: 1.364242e-08 error= 2.07 R method: 4.444444e-09 error= 0 Expected root: 1.111111e-09 Naive method: 0 error= 1 R method: 1.111111e-09 error= 0
triroot <- function(a, b, c) { b <- b/2 d1 <- b**2 d2 <- a*c if(isTRUE(all.equal(d1,d2))) { return(-b/a) } else if(d1 < d2) { return(NULL) } else { h <- -(b + sign(b)*sqrt(d1-d2)) return(c(c/h, h/a)) } }
TODO Autour du dépassement de capacité
STARTED Extension aux racines complexes
Vous avez de la chance, il existe une classe complex d’emblée intégrée à R, donc sans import.
- Cherchez dans la documentation ou sur le web comment définir les nombres complexes \(z_1=3-2i\), \(z_2=5+i\) et \(z_3=i\).
- Calculez l’addition \(z_1 + z_2\), le produit \(z_1 \times z_2\) et l’inverse $\frac{1}{z_3}. Vérifiez que \(z_3\) est bien la racine carrée de -1 …
- Reprogrammez la méthode
trirootretournera aussi les racines complexes. - Calculez les racines des polynômes \(2x^2 - 3x -2\), \(x^2+x+1\), et \(x^2+1\).
z1 <- 3-2i z2 <- 5+1i z3 <- 1i z1 + z2 z1 * z2 1/z3
[1] 8-1i [1] 17-7i [1] 0-1i